分類などの機械学習モデルを構築するときにデータセットに偏り(不均衡データ)があると適切に学習できない可能性がある.データセットを強制的に増やす操作を「オーバーサンプリング」と言って,SMOTE (Synthetic Minority Over-sampling Technique) や ADASYN (Adaptive Synthetic) など,具体的な「オーバーサンプリング手法」がよく知られている.ちなみに SMOTE は k-NN (k-Nearest Neighbor) : k近傍法 を参考に近接データを増やす.
imbalanced-learn とは
今回紹介する imbalanced-learn は「不均衡データ」を扱うライブラリで「オーバーサンプリング」や「アンダーサンプリング」などを簡単に実装できる.そして scikit-learn と互換がある.また GitHub だと scikit-learn-contrib プロジェクトで管理されている.
SMOTE モジュールを試す
今回は imbalanced-learn に入門するために SMOTE モジュールを試す.Over-sampling のドキュメントに載っているサンプルコードを参考にしつつ,もっと簡単に書き直してみた.
- 2. Over-sampling — Version 0.8.1
- SMOTE — Version 0.8.1
- sklearn.datasets.make_classification — scikit-learn 1.0.1 documentation
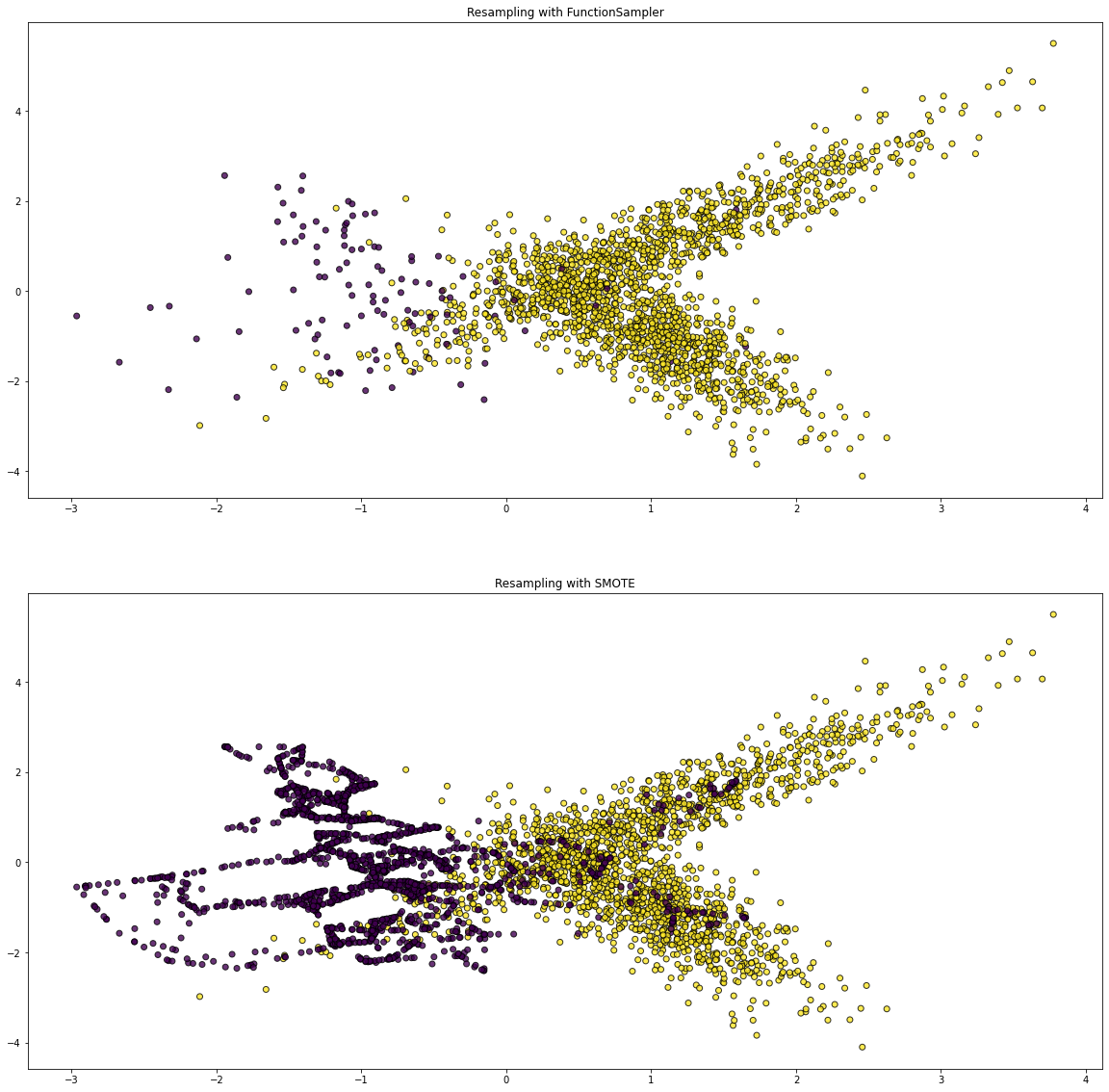
まず,scikit-learn の make_classification() 関数を使って,2000 データセットを 0.05 : 0.95 の割合で不均衡データとして生成した.特徴量はシンプルに2個にした.そして imbalanced-learn の SMOTE モジュールで fit_resample() 関数を使うと簡単に「オーバーサンプリング」をすることができる.
from imblearn import FunctionSampler from imblearn.over_sampling import SMOTE from sklearn.datasets import make_classification import matplotlib.pyplot as plt def plot_resampling(X, y, sampler, ax): X_res, y_res = sampler.fit_resample(X, y) ax.scatter(X_res[:, 0], X_res[:, 1], c=y_res, alpha=0.8, edgecolor="k") title = f"Resampling with {sampler.__class__.__name__}" ax.set_title(title) X, y = make_classification( n_samples=2000, n_features=2, n_informative=2, n_redundant=0, n_classes=2, weights=[0.05, 0.95], class_sep=1.0, random_state=1 ) fig, axs = plt.subplots(nrows=2, ncols=1, figsize=(20, 20)) samplers = [ FunctionSampler(), SMOTE() ] for ax, sampler in zip(axs.ravel(), samplers): plot_resampling(X, y, sampler, ax)
そして matplotlib の scatter() 関数で散布図を描画した.Resampling with FunctionSampler(通常) と Resampling with SMOTE(オーバーサンプリング) を比較すると,特徴量(紫色)に大きく差を確認できる.便利だ!